서 론
기관지천식은 전 인구의 10% 이상에서 관찰될 정도로 흔한 만성 기도 질환으로 국내에서도 산업화 및 환경 변화로 인해 최근 꾸준히 증가하고 있다. 천식은 호산구 침윤을 특징으로 하는 기도 염증 소견, 기관지 과민증 및 기도의 가역적 폐쇄 등이 특징으로 한다.
천식의 유전적 배경은 1990년대 초부터 가계도 연구 및 쌍둥이 연구를 통해 36% 이상으로 추정되었다. 즉, 천식의 발생에 환경적 인자뿐 아니라 유전적 인자의 중요성이 부각되었다. 그 이후 염기서열 분석 방법의 기술적 도약에 발맞춰 천식의 유전적 소인을 규명하고자 하는 유전체 연구가 활발히 진행되면서, 천식 관련 유전자 좌(locus)의 규명뿐 아니라 기도과민증, IgE치의 증가, 아토피와 같은 천식의 주요 표현형과 관련된 유전자 변이들이 보고되었다.
본 종설은 기관지천식의 유전적 소인을 규명하고자 하는 유전체 연구 현황을 시대별로 살펴보고 그 연구결과로 밝혀진 기관지천식의 주요 유전적 소인들을 소개하고자 한다.
1. 천식의 유전적 요소
초기 연구들을 살펴 보면, 천식의 유전적 성향은 가계도 연구 및 쌍둥이 연구를 통해 36~79%로 추정되었다1). 1994년 Lawrence 등2)은 영국인 131 가구를 대상으로 설문지를 토대로 한 천식 지표와 분리 분석(segregation analysis) 연구 결과 28% 유전률(heritability)을 관찰하였고, 1997년 Jenkins 등3)도 7,394 가계도 분리 분석 연구 결과에서 15.9% 유전률을 보고하였다. 한편 1971년 6,996명의 쌍둥이를 대상으로 한 대규모 쌍둥이 연구(스웨덴)에서 일란성 쌍둥이의 경우 19% 일치율(concordance rate)과 이란성 쌍둥이의 경우 4.8% 일치율을 보고하였다4). 또한 13,888명의 핀란드 쌍둥이 연구에서도 일란성 쌍둥이에서 13%, 이란성 쌍둥이에서 7%의 일치율이 관찰되었고, 다중분석 결과 천식의 유전률은 36%로 추정되었다5). 3,808명의 호주 쌍둥이 연구에서는 일란성 쌍둥이의 경우 65%, 이란성 쌍둥이의 경우 24%를 보였고, 유전률은여성에서 60%, 남성에서 75%를 보였다6). 즉, 천식의 유전률은 인종 집단간에 다소 차이를 보이지만 천식의 발현에 있어서 환경적 인자뿐 아니라 유전적 인자의 중요성이 입증되었다.
그 이후 천식의 유전적 소인을 밝히고자 하는 유전체 연구가 염기서열 분석 방법의 기술적 도약에 발맞춰 1990년대 후반에 가속화 되었다.
2. 천식의 유전체 연구
천식은 외부 항원인 알레르겐에 대한 제 I형 과민 반응과 Th2 면역 반응으로, 1970년대 천식의 병태생리에 중요한 물질인 IgE 항체가 발견되고 동시에 알레르겐에 대한 면역반응에서 조직 적합성 항원(human leukocyte antigen, HLA)의 역할이 알려졌다. 따라서 초기의 유전체 연구는 원인 알레르겐에 대한 면역반응에 의한 천식과 특정 HLA형과의 연관성 연구와 알레르기성 천식 환자에서 특정 HLA형과 혈청내 IgE 치와의 연관성이 보고되었다7).
1980년대 말로 접어들면서 다양한 다형성 유전자 DNA 마커가 보고되었고, 그 중 microsatellite 마커는 유전자 지도 작성과 유전성질환의 해석에 있어, 유용한 최적의 유전자 마커로 각광받게 되었다. 천식의 유전적 연구에 microsatellite 마커와 같은 다형성 DNA 마커들이 활용되면서, 이를 계기로 천식의 유전체 연구도 새로운 전기를 맞이하게 되었다. 1980년대 말에서 1990년대 초 천식의 가계도 연구는 주요 염증 매개체로 작용하는 Th2 사이토카인이 밀접한 염색체 5번8)과 IgE 수용체가 존재하는 염색체 11번9,10)을 타겟으로 여러 개의 microsatellite 마커들을 분석하고 천식의 표현형인 IgE와의 연관성을 분석함으로서 천식 관련 유전자를 규명하였다.
1996년을 기점으로 게놈 연계 분석(linkage analysis) 연구는 종래의 타겟 연구기법에서 벗어나 전체 게놈상 산재되어 있는 수백 개의 microsatellite 마커로 전체 게놈을 스캔하여 천식 관련 유전자가 위치한 염색체를 찾고, 이어서 그 염색체 상에 위치한 여러 개의 유전자내 유전자변이들을 타이핑하는 전장 게놈 연계 분석(genome-wide linkage analysis)으로전환되었다11). 우선, 천식 환자의 가계도내 천식 관련 표현형의 유전 방식과 동일하게 유전되는 microsatellite 마커를 확인함으로써 천식 표현형과 연결된 염색체 내의 유전자좌를 확인하고, 해당 염색체내 유전자를 미세지도분석(fine mapping)하여 천식과 연결된 새로운 유전자를 규명하는 방식으로 진행되었다.
천식 환자에서 가계도 기반 게놈 연계 분석 결과, 인간 염색체 2q22-33, 5q23-31, 5p15, 6p21.3-23, 11p13, 11p15, 12q14-24.2, 13q21.3, 14q11.2-13, 17p11.1-q11.2, 19q13, 21q21 등이 천식 및 중간 표현형과 연결된 유전자 좌로 선정되었다12,14). 이 중 가장 일관되게 연관성을 보이는 유전자 좌는 IL-3, IL-4,IL-5, IL-9, IL-13, IFNG, FCER1B 등과 같은 유전자들이 포함된 5q23-31, 5p15, 12q14-24.2, 11q13 등이다. 이들은 대부분 Th1 또는 Th2로의 분화에 영향을 주는 유전자들로 호산구, 호중구, 비만세포와 같은 염증 세포들의 기관지 유입에 중요한 역할을 한다.
또한 전장 게놈 연계분석으로 밝혀진 천식 관련 유전자좌를 미세지도분석(fine mapping)하여 천식의 중간 표현형과 관련된 새로운 유전자들이 규명되었다(표 1). 2002년 Van Eerdewegh 등15)은 미국과 영국의 천식 질환을 가진 460가구를 대상으로 게놈 연계 분석을 수행하여 염색체 20번의 유전자좌(20p13)가 기도의 과민반응성과 연관성이 있음을 확인하였고, 해당 유전자 좌의 미세지도분석을 수행하여 ADAM33(a disintegrin and metalloproteinase 33)이라는 새로운 유전자가 기도과민성에 기여하는 주요 유전적 소인이 됨을 보고하였다. 이 연구를 기점으로 DPP10 (dipeptidyl-peptidase 10)16), PHF11 (plant homeodomain finger protein 11)17), GPRA (G-protein-coupled receptor for asthma susceptibility)18), HLA-G19), CYF1P2 (fragile X mental retardation protein interacting protein 2)20), IRAKM (IL-1 receptor-associated kinase-M)21), 그리고 OPN322) 유전자들이 해마다 발표되었고, 이들은 천식의 새로운 유전적 소인으로 각광을 받았다. 이후 해당 유전자의 유전자 변이에 따른 기능 변화 연구들이 시도되어 어떤 분자 기작을 통해 기관지천식의 표현형에 기여하는지에 대한 연구가 지속적으로 발표되고 있다. 전체 게놈 연계 분석은 가계도 연구를 통해 질환 및 중간 표현형과 연결된 염색체 내의 유전자좌를 확인하는 것으로 기존의 어떤 가설도 기초로 하지 않아 새로운 유전자를 발굴 할 수 있는 장점이 있다. 그러나 질환 가계 대상 확보의 어려움과 고가의 분석비용 등 연구의 제한 점이 있다.
1996년을 기점으로 DNA 염기서열 결정 방식에 초고속스캔 기법이 도입되었고 인간 게놈 프로젝트의 수행으로 인간 게놈 내 수백만개 이상의 단일 염기 다형성들의 존재가 밝혀졌다. 이에 다수의 연구자들은 단일 염기 다형성과 같은 유전자 다형성과 천식의 중간 표현형과의 연관성을 규명하는 유전자 연관성 연구(genetic association study)로 방향을 전환하였다. 우선, 천식의 병태 생리를 바탕으로 후보 유전자를 선정하고, 유전자 내 유전자 변이를 조사한 뒤, 해당 유전자 변이형 빈도를 천식군과 정상 대조군에서 비교 분석함으로써 천식 관련 유전자 변이를 발굴하였다. 지금까지 보고된 천 편 이상의 천식 관련 연관성 연구 결과를 토대로 구축된 NCBI의 유전자형-표현형 데이터베이스(dbGaP, http://www.ncbi. nlm.nih.gov/projects/gap)에서 천식과 관련 있는 유전자를 검색해 보면 총 540개의 천식 관련 유전자의 탐색이 가능하다. 천식 유전체 연구 결과는 2008년 Vercelli D23) 등에 의해 천식의 병태생리 및 병인기전을 기초로 다음과 같이 4개로 분류되었다23). 1) 선천성 면역반응(innate immunity)과 면역조절 반응에 관여하는 유전자, 2) Th2 세포의 분화 및 효능 작용 관련 유전자, 3) 기도 상피세포 관련 유전자, 4) 폐기능 및 질병 중증도 관련 유전자 등이다. 주요 유전자들을 각각 살펴 보면, 염색체 6번에 위치한 HLA은 선천성 면역반응에 중요한 유전자로 특정 알레르겐을 염증세포에 제시함으로써 알레르겐 특이 염증반응을 유도한다. HLA 유전자를 고해상도 조직적합성항원 분석법과 일배체(haplotype) 분석법을 통해 확인한 결과, DRB1, DPB1, DQB1 대립유전자가 천식의 중간표현형과 연관성이 있었다. IL4R (Interleukin-4 receptor) 유전자는 사이토카인 밀집지역인 염색체 5번에 위치하면서 Th2 사이토카인 IL4와 IL13의 수용체로 IgE 생성 및 염증 반응에 기여한다. 기도 상피세포에서 분비되는 eotaxin, RANTES와 같은 케모카인(chemokine)은 화학주성이 있어 염증세포를 기도 내로 유입되도록 유도하여 염증반응을 지속시킨다. ADRB2 (β2-adrenoceptor) 유전자는 천식 치료제인 베타2 항진제 수용체로 천식의 표현형, 중간 표현형뿐 아니라 베타 항진제에 대한 약물반응성에도 관여함으로 향후 활발한 연구 결과가 기대된다.
그 이후 2006년도를 기점으로 천식 관련 유전체 연구는 새로운 국면을 맞이하게 되었다. 인간 게놈 프로젝트, 단일염기다형성 프로젝트, 일배체형 프로젝트를 진행하면서 수 백만개 이상의 단일 염기 다형성들이 보고되었고, 이 방대한 정보를 기초로 50만개 단일 염기 다형성 분석이 동시에 가능한 단일 염기 다형성 어레이(SNP array)가 사용화되었다. 따라서 단일 염기 다형성 어레이를 이용한 전장 게놈 연관성 연구(whole genome-wide association study)가 천식 환자에서 시도되었다. 마침내 2007년 Moffat 등24)이 994명의 소아 천식과 1,243명의 정상 대조군을 대상으로 전체 게놈 연관성 연구를 수행하여 ORMDL3라는 새로운 유전자가 소아 천식의 주요 유전적 소인이 됨을 규명하였다. 그 이후 SNP 어레이를 이용한 전장 게놈 연관성 연구가 활발히 진행되고 있으며 지금까지 보고된 결과는 표 3에 요약하였다25-29).
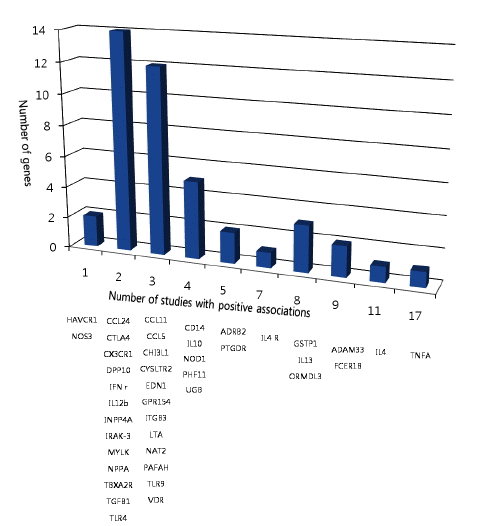
단일 염기 다형성을 이용한 유전자 연관성 연구는 천식환자군과 정상 대조군을 대상으로 진행하여 질환 대상 확보가 상대적으로 용이하고 후보 유전자를 타겟하여 진행하므로 합리적인 분석비용으로 연구가 가능하다. 그러나 특정 집단 내 유전자 연관성이 다른 집단 내에서 재현성을 보이지 않는 경우도 비일비재하여 다른 집단에서의 재현 연구가 필수적이다. 그림 1은 천식 표현형과 관련된 유전자 연관성 연구 결과에서 높은 재현성을 보이는 유전자들을 소개하고 있다. TNFA 유전자는 염증의 증폭에 매우 중요한 신호를 전달하여 천식의 병인 기전에 중요한 역할을 담당하는 것으로 보고되고 있으며 여러 집단내 유전자 연관성 연구 결과 기관지천식의 주요 유전적 소인으로 보고되었다. Th2 염증 반응에 중요한 역할을 담당하는 IL4, IL13, IL4R 유전자들 역시 천식과의 연관성 보고에서 높은 재현성을 보이고 있다. 또한 전장 게놈 연계 분석으로 규명된 ADAM33 유전자와 전장 게놈 연관성 연구로 새롭게 규명된 ORMDL3 유전자 역시 다른 집단 내 높은 유의성을 나타내어 기관지천식의 주요 유전적 소인으로 작용하는 것이 밝혀졌다.
3. cDNA 마이크로 어레이를 이용한 기관지천식 유전자 프로필 조사
천식 환자의 폐생검, 알레르겐 유발 검사 후 폐세척액 또는 단핵구 세포를 대상으로 마이크로 어레이를 수행하여 천식에서의 유전자 프로필을 조사함으로써 유전적 소인을 찾고자 하는 시도가 제한적으로 수행되었다.
2005년 Lilly 등30)은 알레르겐 기관지유발시험 전후 기도상피세포에서의 mRNA 발현 프로필을 비교한 결과 총 141개의 과발현 유전자와 8개의 저발현 유전자를 발견하였고, 이 중 IL-3, IL-4, IL-13, IL-5RA, NFkB와 같은 염증 관련 유전자들이 알레르겐 유발 후 과발현됨을 확인하였다. 2004년 Laprise 등31)은 4명의 천식 환자들과 4명의 정상인들을 대상으로 기관지 생검에서의 유전자 프로필을 비교 분석하였고, 79개의 유의한 유전자들을 발견하였으며 CX3CR1 (Fractalkine receptor)를 새로운 천식 관련 유전자로 규명하였다. 2005년 Guajardo 등32)은 소아 천식 환자의 비강 점막내 상피 세포에서의 유전자 프로필을 분석하였고, 면역 반응 관련 유전자들의 발현이 천식 악화시 증가되는 가운데 SOCS-3 (suppressor of cytokine signaling 3)와 ITGA4 (Integrin alpha 4) 유전자의 발현이 증가됨을 보고하였다. 2009년 Aoki 등33)은 소아 천식 환자의 말초단핵구 세포에서 유전자 프로필을 조사하였고, RBP7 (retino binding protein 7) 유전자가 천식 악화시 발현이 증가됨을 보고하였다. 그러나 이와 같은 폐생검, 폐상피세포 또는 단핵구 세포를 대상으로 유전자 프로필을 조사함에 있어서 어려운 점은 세포의 이질성(Heterogeneity)이다. 특정 유전자의 발현 차이가 이질적인 세포의 오염으로부터 기인할 수 있기 때문이다. 또한 마이크로 어레이에서 관찰된 유전자 발현의 차이는 이후 면역화학적 방법 또는 In situ hybridization 같은 방법으로 유전자 발현을 확인할 필요가 있다. 최근 LCM (lase capture microdissection)이라는 방법이 도입되어 세포의 이질성 문제는 극복 가능하나 인간 생검을 대상으로 수행하는데 있어서 여러 제약이 따른다.
결론 및 향후 전망
천식 유전체 연구는 염기서열 분석 기법의 발전과 맞물려 지속적으로 진화되고 있다. 최근, 인간 염기서열 분석 기법의 놀라운 발전으로 보다 빠르게 보다 정확하게 인간 전체 게놈의 염기서열 분석이 가능하게 되었다. 마침내, 영국, 미국, 중국 등의 국제공동 연구팀은 일배체형 프로젝트에 참여한 1,000명의 지원자들의 게놈을 분석하여 3,199가지의 인간 질병을 야기하는 유전적 변이를 조사 분석하여 질병관련 유전자 변이 프로필을 만들고자 하는 제2차 게놈 프로젝트인 1,000 게놈 프로젝트가 2008년 시작되어 2011년 완성 예정이다.
또한 유전체 연구분야뿐 아니라 단백체, 대사체, 후생유전학 등의 연구기법도 발전을 거듭하고 있어 기관지천식과 관련된 유전적 소인의 규명뿐 아니라 천식 관련 기전 연구에 박차를 가하게 될 것이다.
천식의 유전적 소인을 밝히기 위한 연구는 cDNA 마이크로 어레이를 이용한 유전자 발현 차이 조사 연구와 단일 염기 다형성을 이용한 유전자 연관성 연구로 대변할 수 있다. 그 연구 결과를 토대로 지금까지 많은 유전자들이 천식 표현형과의 연관성이 보고되고 있으며 해당 유전자들의 기능 연구를 통해 분자 수준의 기전을 밝힘으로서 천식의 병인기전을 이해하고 새로운 치료제를 개발하는데 응용하고자 한다.
향후 더 많은 연구결과가 축적되면, 유전자 정보에 근거하여, 천식과 약물 알레르기 환자의 표현형을 예측하기 위한 진단법 개발과 치료제에 대한 반응 및 약물 이상반응을 예측하여 개인에 따른 맞춤치료를 적용할 수 있는 시대가 오리라 기대한다.



 PDF Links
PDF Links PubReader
PubReader ePub Link
ePub Link Full text via DOI
Full text via DOI Download Citation
Download Citation Print
Print