후성유전과 알레르기 질환
Epigenetics in Allergic Diseases
Article information
Trans Abstract
The epigenetics is the study of changes in gene expression or celluar phenotype caused by mechanisms other than changes in the underlying DNA sequence. Epigenetic regulation in asthma and allergic diseases is an interesting area that has obtained a great deal of scientific concern in recent years. Allergic diseases such as asthma and allergic rhinitis in early life are one of the important indicators that the developing immune system is vulnerable to biologic and environmental pollutants. The epigenetic studies are likely to provide clear and deep understandings into how the environment can be modified to drive immune development and change the allergic disease phenotypes. This review includes 1) definition of epigenetics 2) regulation of epigenetics 3) characterization of environmental asthma and allergic diseases triggers that induce epigenetic changes. (Korean J Med 2013;85:260-266)
서 론
천식은 여러 세포와 다양한 매체들이 관여하는 만성 염증성 기도 질환이다. 기도의 염증은 기도과민증과 반복적인 천명, 호흡곤란, 가슴 답답함, 기침 등의 증상을 일으킨다. 천식의 위험인자(Table 1)는 천식을 발생시키는 원인 인자와 증상을 일으키는 유발 인자 두 가지로 나눌 수 있다. 이러한 인자가 천식의 발생과 증상 발현에 복잡한 상호 작용을 한다.
후성유전학(epigenetics, 後成遺傳學)은 DNA 염기서열의 변화 없이 세포 기능이나 개체의 형질변화에 관여하는 메커니즘을 연구하는 학문이며 이러한 현상을 전 유전체를 대상으로 연구하는 것이 후성유전체학이다[1]. 최근 후성유전 조절은 세포 분화를 포함한 다양한 조절반응을 이해할 수 있는 하나의 분자 메커니즘으로 세포의 기능 분석 및 질환의 병인 규명 연구에 이용되고 있다. DNA 메틸화, 히스톤 tail의 변화(메틸화/탈아세틸화), Non-coding RNA 등으로 연구되고 있고 이러한 후성유전의 변화에 환경, 음식, 질병, 나이 등이 영향을 미치는 것으로 알려져 있다[1-3]. 이 논고에서는 후성유전학을 소개하고 알레르기 질환에서 후성유전이 미치는 영향에 대해 소개하고자 한다.
후성유전학
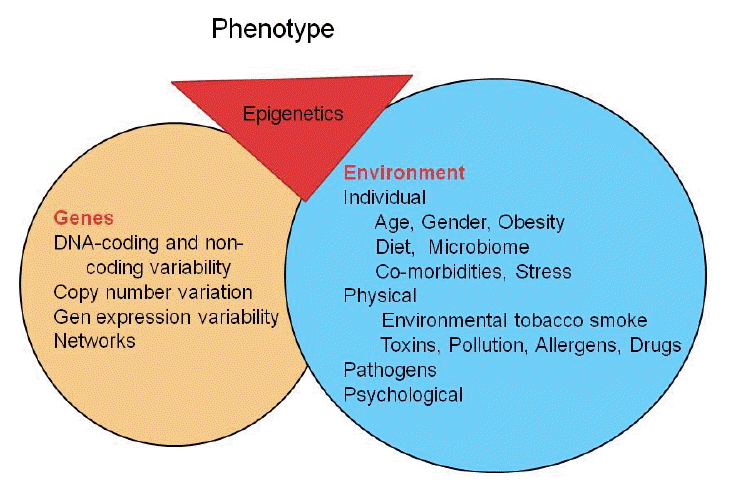
후성유전학(Fig. 1)은 DNA 염기서열의 변화 없이도 유전자 발현 패턴 및 유전자 발현의 활성이 변화되고 이것이 다음 세대로 유전되는 현상을 연구하는 학문분야이며 DNA 메틸화, 포스포릴화, 유비퀴틸화, 히스톤 tail의 변화, Non-coding RNA 등 여러 가지 형태가 있으며 이들 중 DNA 메틸화 연구가 가장 잘 알려져 있다[4-6].
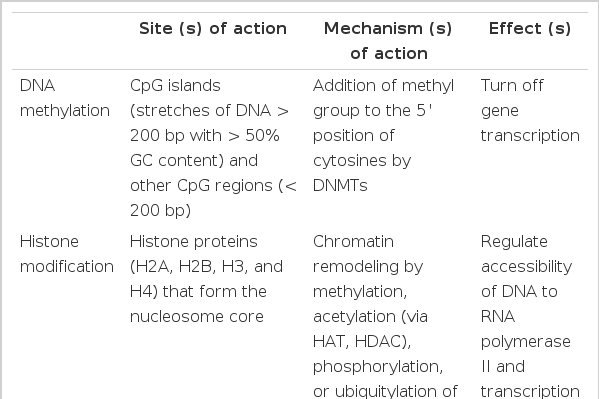
유전자와 환경인자의 상호작용을 통해 천식의 감수성을 결정하며 이러한 환경과 유전자의 발현에 후성유전이 영향을 준다. Aberrant DNA methylation, histone modification, 특이 microRNA 발현, chromatin 변화 등이 면역반응에서 T-세포반응, 돌기세포기능, 탐식세포 활성화, 기도 상피세포 장벽 등 천식에 영향을 미치고 후기 장년에 중증도를 증가시킨다[7-30]. 유전자 전사과정에 대한 후성학적 조절 기전(Table 2)은 1) DNA 메틸화 2) 히스톤 메틸화/탈아세틸화 3) Non-coding RNA 등 서로 다른 세 가지 기전으로 연구되고 있다.
유전학적으로 돌연변에 의한 염기서열 변화가 중요한데 후성학적으로는 시토신(C)의 피리미딘 고리 C5 자리에 메틸기가 붙는 메틸화에 의해서 유전자의 발현이 변하고 이 반응은 시토신(C) 뒤에 구아닌(G)이 올 때에만(CpG) 일어나고 DNA 메틸기전달효소(DNA methyltransferase, DNMT)에 의해서 매개되며 S-adenosyl-methionine이 메틸기로 작용한다. 이 배열에서 시토신(cytosine)이 메틸화되는 경향이 많아 인간게놈의 경우 전체 시토신 중 3-5%는 메틸화(5-methylcytosine)되어 있다.
CpG는 인간이 진화과정에서 점차 감소되어 왔으며 genome에 존재하는 CpG의 메틸화 정도와 양상은 포유동물의 종에 따라 다르고 조직에 따라서도 다른 양상을 보인다. 포유동물의 염기서열에는 CpG가 밀집되어 있는 CpG island (stretch of DNA > 200 bp in length with > 50% GC content, observed/expected CpG > 0.6) [31]라는 부위가 존재하며 이 부위는 0.5-4 kb 정도로 게놈의 유전자와 밀접한 연관성을 가지고 있는 것으로 생각한다. 이 CpG island는 유전자의 전사과정을 조절하는 프로모터 부근에 위치하지만 대부분의 CpG island는 메틸화되지 않는다. 따라서 특정 유전자의 발현을 개시하는 프로모터 부위의 CpG island가 메틸화되어 있으면 그 특정 유전자의 발현이 억제되고 메틸화가 되지 않으면 유전자의 발현이 진행한다.
DNA 메틸화는 메틸기 전달효소에 의해 일어나는데 DNMT1은 세포분열 과정에서 DNA가 합성될 때 DNA 메틸화 상태를 유지시키는 기능을 지닌다. DNMT3a와 DNMT3b는 새로운 메틸화를 촉매하는 기능을 갖고 있는 것으로 여겨지고 있다. DNA 메틸화 자체가 단순히 전사인자의 결합을 방해함으로써 유전자 전사를 억제하기도 하고 여러 종류의 전사 인자들이 자신의 인식 부위에 위치하는 메틸화 CpG에 민감한 것으로 알려져 있다. 메틸화는 전사인자의 인식을 방해하며 일단 DNA가 메틸화되면 이 부위에서 메틸기 결합(Methyl-Binding Domain, MBD) 단백질이 유도된다. 이 단백질 중 MeCP2, MBD2, MBD3는 히스톤 탈아세틸효소 등과 결합하여 복합체를 형성한다. 유전자의 메틸화는 메틸기 전달효소의 활성보다 히스톤을 변형 또는 염색질 리모델링에 의해 전사과정을 억제하고 있다.
뉴클레오솜은 DNA 약 146쌍 정도와 4종류의 핵심 히스톤들(H2A, H2B, H3, H4)로 이루어져 있다. 히스톤은 구형의 본체와 아미노 꼬리 부위를 가지고 있다. 히스톤 3에 있는 리신4라는 특정 리신 잔기에 메틸화(H3K4me3)가 일어날 때 유전자가 활성화되고 히스톤 3에 있는 리신27이라는 특정 리신 잔기에 메틸화(H3K27me3)가 일어날 때 유전자 발현이 억제된다[1]. 히스톤 acetly transferase (HATs)는 히스톤 아미노꼬리를 아세틸화하고 히스톤 deacetlylases (HDACs)는 히스톤 아미노 꼬리에서 아세틸 그룹을 제거한다.
MicroRNAs (miRNAs)는 약 22개의 뉴클레오타이로 구성된 RNA이며 인간게놈에서 1,000개 이상이 알려져 있으며 유전자의 발현에 영향을 미치는 것으로 알려져 있다[31-35].
알레르기 질환에서의 역할
천식관련 유전자
천식 환자의 기관지 점막에서 HAT/HDAC 변화가 있으며 HDAC1과 HDAC2의 발현이 감소하고 HAT 활성 증가하며[21] 천식 어린이 말초혈액 단핵구에서 HAT/HDAC 비율이 변하고 HDAC 발현 감소 HAT성도 증가가 관찰되며 기도과민성과 관련이 있었다[36]. 천식유전자 중 IL-4, IL-5, IL-13, TGFβ, IFNγ, HLAG, FOXP3+ 등 여러 유전자가 후성유전에 의해 조절되는 보고들이 있다[5-9]. 알러젠 노출은 Th세포를 Th2로 분화시키고 알레르기 반응을 일으키는 IL-4, IL-5, IL-13를 분비한다. DNA 메칠화가 안되거나 히스톤 활성화는 Th2세포의 Th2 locus에 euchromatin을 유지하여 Th2 사이토카인의 발현을 증가시킨다. GATA 3유전자 인트론의 전사 부위에 CpG 메틸화가 결손되고 IL-4 locus에 histone 3-lysine 9 아세틸화 및 histone 3-lysine 4의 트리메틸화로 IL-4 발현이 증가하고 IFN-γ가 감소한다[37-40]. 그리고 Th1 분화는 IL-4 locus에 메틸화와 관련되며[38] IFN-γ promoter 부위의 activator protein 1-binding site의 CpG 메틸화는 cAMP-responsive element binding protein 1 (CREB)과 activating transcription factor 2(ATF2)/c-Jun 결합이 cis-regulatory element 결합을 억제하여 유전자 발현을 막는다[41,42]. 이와 같이 다양한 유전자의 DNA 메틸화, 히스톤 tail의 변화(메틸화/탈아세틸화), miRNAs 등을 통해 알레르기 염증이 변화에 관여한다(Table 3).
환경인자
흡연, 대기오염, 지질 다당류, 음식, 알러젠 등 다양한 환경적인 요인들이 후성유전을 통해 천식에 영향을 미친다. 흡연은 천식에 중요한 위험인자이고 사람이나 동물모델에서 흡연에 노출되면 알러젠 감작을 증가시키고 초기에 흡연 노출은 후기 천식 발생을 증가 시킨다[43]. 흡연은 직ㆍ간접적으로 산화 스트레스를 통해 후성유전에 영향을 미친다. 흡연자는 기도에서 기관지 내시경과 기관지 폐포세척액을 이용하여 비흡연 성인과 비교하였을 때 HDAC2 발현과 HDAC활성도가 감소하고 HAT/HDAC 균형이 깨지며 GM-CSF, IL-8, IL-1β-induced TNF-α 등이 증가한다[44]. 흡연이 탐식세포에서 스테로이드 저항성을 가져오는데 HDAC 억제제를 처리하면 흡연에 의한 염증을 줄여서 치료 측면에서도 효과적일 수 있다[45]. 또한 흡연은 p16 (cyclin-dependent kinase inhibitor 2A [melanoma, p16, inhibits CDK4; INK4a]), cytochrome P450, family 1, subfamily A, polypeptide 1 (CYP1A1) [46], Ras association(RalGDS/AF-6) domain family member 1 (RASSF1A)[47], fragile histidine triad gene (FHIT) [46], monoamine oxidase type B (MAOB) [48] 등의 후생유전변화에 관여한다. 그리고 임신 시 할머니가 흡연할 경우 손자의 천식과 관련을 갖고 환경요인이 세대에 걸쳐 영향을 미치는 것으로 알려져 있다[49].
대기오염 물질은 건강에 위해한 영향을 미치고 있어 천식 및 만성 폐쇄성 폐질환 등 호흡기 질환이 있는 사람에서 폐기능 저하, 기도 과민성 증가, 기도 염증을 유발하고 대기오염이 증가될 때 천식환자가 응급실 내원, 병원 입원, 약물 사용 등이 증가되는 것으로 알려져 있다.
디젤분진 노출 생쥐에서 IgE 증가, IFNγ promoter의 CpG(245), CpG (253)와 CpG (2205) 위치에 과메틸화, IL4 promoter의 CpG (2408)에 저메틸화를 가져온다[50]. 디젤분진, 벤조 피렌, PM-10은 산화 스트레스로 histone 아세틸화, 크로마틴 재개형과 사이토카인 증가를 가져온다[51].
임신 시 메틸기 음식을 먹은 산모 동물에서 림프구를 Th2분화하고 태어난 쥐에서 여러 가지 유전자(134, 82-gene loci)에서 메틸변화가 관찰되고 임신 중에 메틸 donor로 folate, vitamin B12, choline, methionine, genistein, betaine 등을 많이 섭취한 새끼 쥐나 흡연 쥐에서 알레르기 기도 질환을 억제하는 것으로 알려진 Runt-related transcription factor 3 (Runx3) 메틸화로 새끼 쥐에서 Runx3 유전자 발현이 감소하여 천식 표현형이 증가한다[52]. 이러한 기전으로 일본 연구에서는 자녀에서 folate나 비타민 D 섭취가 자녀의 천명에 영향이 없었으나 노르웨이 코호트(> 32,000 children)나 오스트리아 연구에서는 엽산을 엄마가 먹었을 때 아이에서 천명이 증가하고 호흡기 감염이 감소한 것으로 보고하였다[53]. 그리고 최근 천식과 비타민 D와의 연관성(54-56)에 대한 연구와도 관련성을 제시하는 근거가 되고 있다.
TLRs는 흡입항원에 선천면역반응에 중요한 역할을 하는데 집먼지 진드기 노출은 TLR4를 증가시키고 miRNAs인 miRNA-16, miRNA-21, miRNA-126 [57]를 증가시키고 miRNA-126 선택적 억제제가 miR-126-mediated GATA-3 발현 억제를 통해 천식증상을 감소시키고 Th2 반응을 감소시키며 기도과민증과 염증을 감소시킨다. 어릴 때 감염은 산소종과 염증중재자의 생성을 증가시키고[58,59] 산소종이 사이토카인의 메틸화 유전자의 발현을 억제하여[60] 천식을 억제하고 후기 감염은 천식의 악화에 관여하는 것으로 알려져 있다[61-63]. 기도에서 산소종과 염증매개물질은 아세칠화/비아세틸화 균형에 영향을 미치며[64,65] HAT/HDAC불균형은 IL-8, IL-6, CXCL1, CXCL2, CXCL3 등의 증가를 가져온다. 세균에서 분비되는 endotoxin에 의해 천식이 억제되거나 천식의 발생 및 억제를 가져오는데 endotoxin tolerance는 IL-1β-mediated TNF-α 분비와 관계되고 endotoxin이나 LPS 노출은 IL1B 프로모터 nucleosome의 크로마틴 재형성과 TNFα 유전자의 발현을 억제한다[66-68].
결 론
후성유전이 천식 및 알레르기 질환에 미치는 영향은 앞으로 많은 연구가 필요하며 이런 연구를 통해서 1) 알레르기를 일으키는 원인인 알러젠, 대기오염, 담배연기, 바이러스 등이 천식에서 후성유전의 변화를 가져올 것인지, 2) 천식의 임상 경과 중 엄마 뱃속에서부터 그 이후로 출생과정, 성장과정 중 어느 과정에 영향을 주는지, 환경요인에 의해 연령에 따라 다른 영향을 주는지, 한 번 생긴 변화가 바뀔 것인지, 3) 후성유전이 유전자와 상호작용은 어떠한지, 4) 후성유전의 변화를 알거나 제어할 수 있는 생체 마커는 무엇인지 등에 대한 연구가 이루어져 천식 및 알레르기 질환에 후생유전이 미치는 영향이 파악되고 이러한 영향을 줄이거나 극복하는 치료제가 나오기를 기대한다.